Анализ колоса пшеницы методами компьютерного зрения. Определение плоидности
14-ого августа завершился первый воркшоп Математического центра в Академгородке. Я выступал в роли куратора проекта по анализу колоса пшеницы методами компьютерного зрения. В этой заметке хочу рассказать, что из этого вышло.
Для генетики пшеницы важной задачей является определение плоидности (число одинаковых наборов хромосом, находящихся в ядре клетки). Классический подход решения этой задачи основан на использовании молекулярно-генетических методов, которые дороги и трудозатратны. Определение типов растений возможно только в лабораторных условиях. Поэтому в данной работе мы проверяем гипотезу: возможно ли определить плоидность пшеницы, используя методы компьютерного зрения, только лишь на основании изображения колоса.

Описание данных
Для решения задачи еще до начала воркшопа был подготовлен набор данных, в котором для каждого вида растения была известна плоидность. Всего в нашем распоряжении оказалось 2344 фотографии гексаплоидов и 1259 тетрапроидов.
Большинство растений фотографировалось по двум протоколам. Первый случай — на столе в одной проекции, второй — на прищепке в 4-х проекциях. На фотографиях всегда присутствовала цветовая палитра колорчекера, она нужна для нормализации цветов и определения масштаба.

Всего 3603 фото с 644 уникальными посевными номерами. В наборе данных представлено 20 видов пшениц: 10 гексаплоидных, 10 тетраплоидных; 496 уникальных генотипов; 10 уникальных вегетаций. Растения были выращены в период с 2015 по 2018 годы в теплицах ИЦиГ СО РАН. Биологический материал предоставлен академиком Николаем Петровичем Гончаровым.
Валидация
Одному растению в нашем наборе данных может соответствовать до 5 фотографий, снятых по разным протоколам и в разных проекциях. Мы разделили данные на 3 стратифицированные подборки: train (обучающая выборка), valid (валидационная выборка) и hold out (отложенная выборка), в соотношениях 60%, 20% и 20% соответственно. При разбиение мы учитывали, что бы все фотографии определенного генотипа всегда оказывались в одной подвыборке. Данная схема валидации использовалась для всех обучаемых моделей.
Пробуем классический CV и ML методы
Первый подход, который мы использовали для решения поставленной задачи основан на существующем алгоритме разработанным нами ранее. Алгоритм из каждого изображения позволяет извлечь фиксированный набор различных количественных признаков. Например, длина колоса, площадь остей и т.д. Подробное описание алгоритма есть в статье Genaev et al., Morphometry of the Wheat Spike by Analyzing 2D Images, 2019. Используя данный алгоритм и методы машинного обучения, нами были обучены несколько моделей для предсказания типов плоидности.
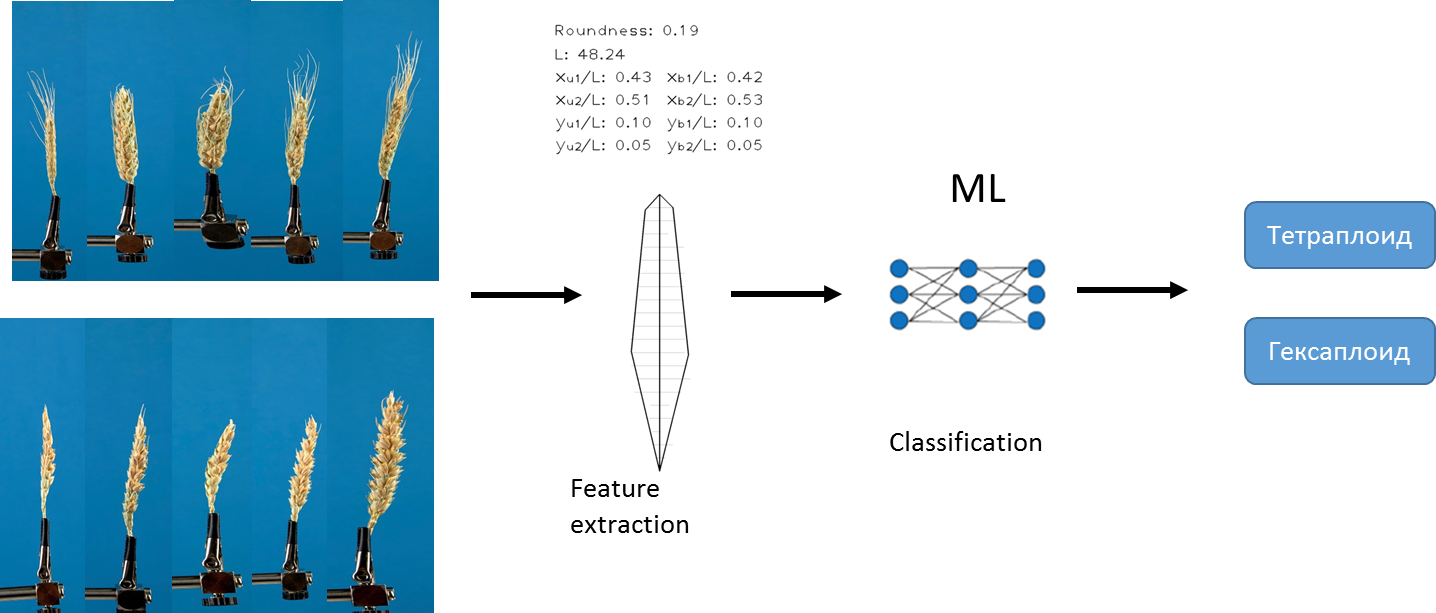
Мы использовали методы логистической регрессии, случайный лес и градиентный бустинг. Данные были предварительно нормированы. В качестве меры точности мы выбрали AUC.
Лучшую точность на отложенной выборке показал метод градиентного бустинга, мы использовали реализацию СatBoost.
Интерпретируем результаты
Для каждой модели мы получали оценку «важности» каждого признака. В результате получили список всех наши признаков, ранжированный по значимости и отобрали топ 10 признаков: Awns area, Circularity index, Roundness, Perimeter, Stem length, xu2, L, xb2, yu2, ybm. (описание каждого признака можно посмотреть тут).
Примером важных признаков являются длина колоса и его периметр. Их визуализация показана на гистограммах. Видно, что распределение для гексаплоидов смещено в сторону больших значений.
Мы выполнили кластеризацию топ 10 признаков методом t-SNE
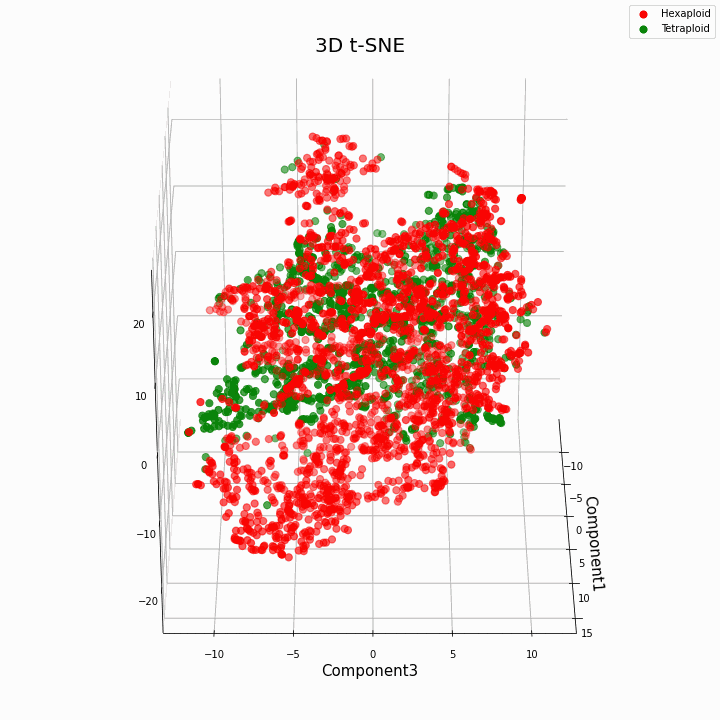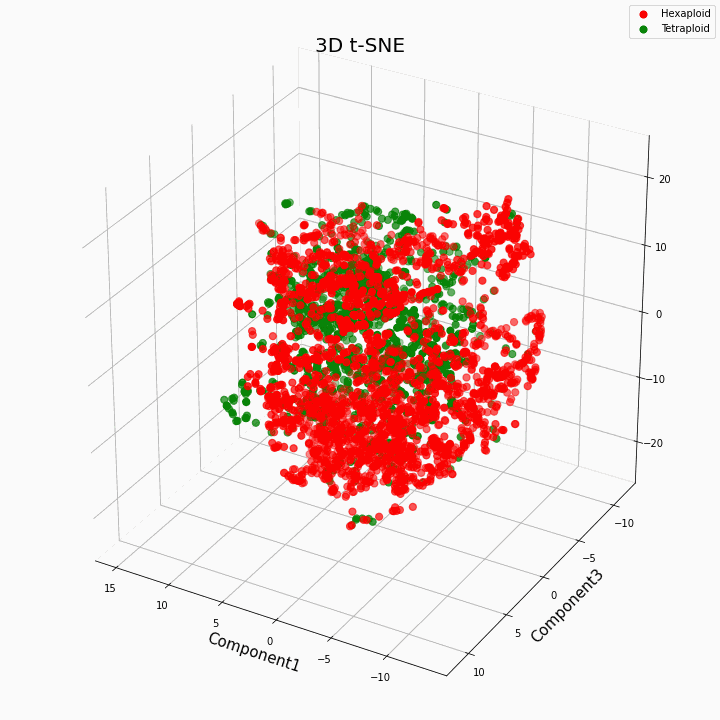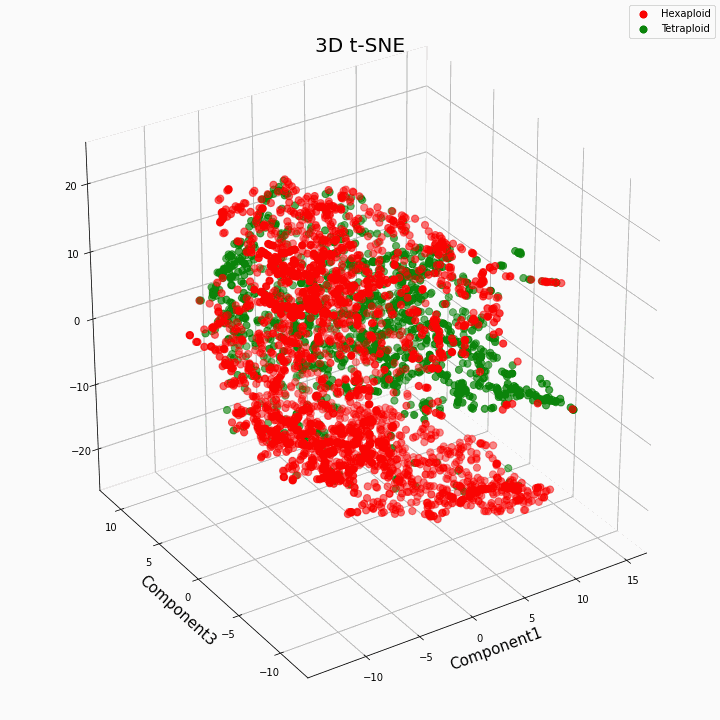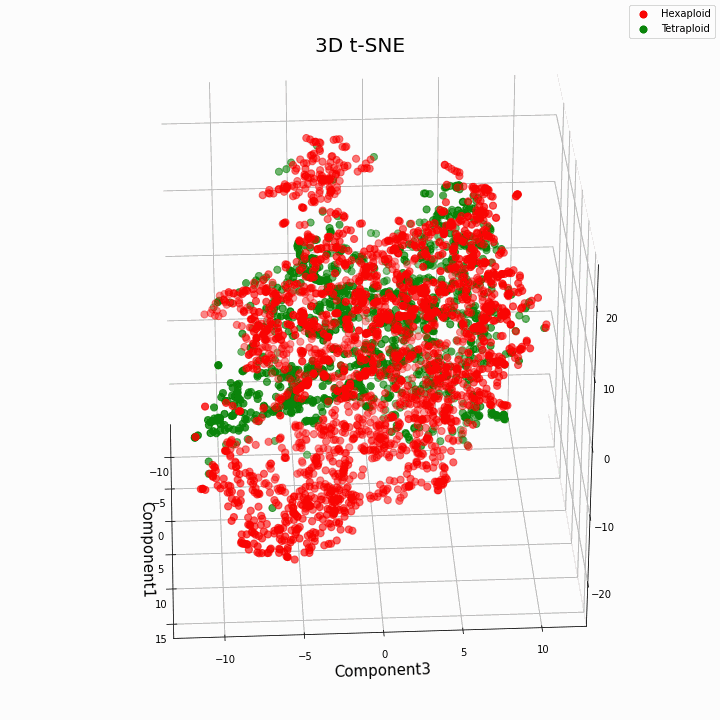
В целом большая плоидность, дает более вариабельные значения признаков. Для гексаплоидов характерна больший разброс/дисперсию значений признака. Это происходит потому что, число копий генов у гексаплоидов больше и поэтому увеличивается число вариантов «работы» этих генов.
Для подтверждения нашей гипотезы о большей фенотипической изменчивости у гексаплоидов мы применили F статистку. F статистика дает значимость различий дисперсий двух распределений. Опровержением нулевой гипотезы о том, что не существует различий между двумя распределениями мы считали случаи, когда значение p-value меньше 0.05. Мы провели этот тест независимо для каждого признака. Условия проверки: должна быть выборка независимых наблюдений (в случае нескольких изображений это не так) и нормальность распределений. Для выполнения этих условий мы проводили тест по одному изображению каждого колоса. Брали, фотографии только в одной проекции по протоколу «на столе». Результаты показаны в таблице. Видно, что дисперсия для гексаплоидов и тетраплоидов имеет значимые различия для 7 признаков. Причем во всех случаях значение дисперсии выше у гексаплоидов. БОльшую фенотипическую вариабельность у гексаплоидов можно объяснить большим числом копий одного гена.
В наших данных представлены растения 20 видов. 10 гексаплоидных пшениц и 10 тетраплоидных.
Мы разукрасили результаты кластеризации так, что цвет+форма каждой точки соответствует определенному виду.
Большинство видов занимает достаточно компактные области на графике. Хотя эти области могут сильно перекрываться с другими. С другой стороны, внутри одного вида могут быть четко выраженные кластеры, например как для T compactum, T petropavlovskyi.
Мы усредняли значения для каждого вида по 10 признакам, получив таблицу 20 на 10. Где каждому из 20 видов соответствует вектор из 10-ти признаков. Для этих данных построили матрицу корреляции и провели иерархический кластерный анализ. Синие квадраты на графике соответствуют тетраплоидам.
На построенном дереве, в общем, виды пшениц разделись на тетраплоидные и гексаплоидные. Гексаплоидные виды четко разделисись на два кластера среднеколосые — T. macha, T.aestivum, T. yunnanense и длинноколосые — T. vavilovii, T. petropavlovskyi, T. spelta. Исключение — к гексаплоидным попал единственный дикий полиплоидный (тетраплоидный) вид T. dicoccoides.
В то время как в тетраплоидные виды попали гексаплоидные пшеницы с компактным типом колоса — T. compactum, T. antiquorum и T. sphaerococcum и рукотворная изогенная линия АНК-23 мягкой пшеницы.
Пробуем CNN

Для решения задачи определения плоидности пшеницы по изображению колоса мы обучили сверточную нейронную сеть архитектуры EfficientNet B0 с предобученными на ImageNet весами. В качестве loss функции использовали CrossEntropyLoss; оптимизатор Adam; размер одного батча 16; изображения ресайзили до разрешения 224×224; скорость обучения меняли, согласно стратегии fit_one_cycle с начальным lr=1e-4. Обучали сеть в течении 10 эпох, накладывая случайным образом следующие аугментации: повороты на -20 +20 градусов, изменение яркости, контрастности, насыщенности, зеркальное отображение. Лучшею модель выбирали по метрике AUC, значение которой считалось в конце каждой эпохи.
В результате точность на отложенной выборке AUC=0.995, что соответствует accuracy_score=0.987 и ошибке равной 1.3%. Что является очень неплохим результатом.
Заключение
Эта работа является удачным примером того, как силами коллектива из 5 студентов и 2-ух кураторов в течении нескольких недель можно решить актуальную биологическую задачу и получить новые научные результаты.
Я хотел бы выразить благодарность всем участникам нашего проекта: Никита Прохошин, Алексей Приходько, Андреевич Евгений, Артем Пронозин, Анна Паулиш, Евгений Комышев, Михаил Генаев.
Ковалью Василию Сергеевичу и Кручининой Юлии Владимировне за съемку колосьев.
Николаю Петровичу Гончарову и Афонникову Дмитрию Аркадьевичу за предоставленный биологический материал и помощь в интерпретации результатов.
Математическому центру Новосибирского Государственного Университета и Институту Цитологии и Генетики СО РАН за организацию мероприятия и вычислительные мощности.
ЗЫ Мы планируем подготовить вторую часть статьи, где расскажем про сегментацию колоса и выделении отдельных колосков.
